Bcftools
Celem przedmiotu jest przekazanie studentom wiedzy i umiejętności niezbędnych do analizy danych z wysokoprzepustowych eksperymentów sekwencjonowania, bcftools.
Slajdy do wykładu są tu i tu. Dane sparowane odczyty z genomu ludzkiego są dostępne tu. Dobrze byłoby zacząć od QC, żeby zobaczyć z czym mamy do czynienia. Potem mapujemy wg. Warto porównać to co wyjdzie nam przy pomocy programu bowtie z opcją -2 i to co wychodzi przy pomocy programu BWA zmapowane odczyty w pliku sam.
Bcftools
.
Będziemy robić zadania z tutorialu, który jest tutaj.
.
In general, whenever multiple VCFs are read simultaneously, they must be indexed and therefore also compressed. BCFtools is designed to work on a stream. It regards an input file "-" as the standard input stdin and outputs to the standard output stdout. Several commands can thus be combined with Unix pipes. This manual page was last updated and refers to bcftools git version 1. See bcftools call for variant calling from the output of the samtools mpileup command. The multiallelic calling model is recommended for most tasks.
Bcftools
In general, whenever multiple VCFs are read simultaneously, they must be indexed and therefore also compressed. Note that files with non-standard index names can be accessed as e. BCFtools is designed to work on a stream. It regards an input file "-" as the standard input stdin and outputs to the standard output stdout. Several commands can thus be combined with Unix pipes. This manual page was last updated BST and refers to bcftools git version 1. See bcftools call for variant calling from the output of the samtools mpileup command. The multiallelic calling model is recommended for most tasks. For a full list of available commands, run bcftools without arguments.
Su stock price tsx
Podobnie jak w p. Spróbuj różnych parametrów preprocessingu flitrowanie trimowanie itp aby zwiekszyc liczbe zmapowanych odczytow. Poza tym chcemy nauczyć się korzystać z pakietu GATK do lokalnego re-alignmentu sekcja 4 i snp-callingu sekcja 5 A także do porównywania znalezionych snpów do dbSNP sekcja 7 przy użyciu podanego pliku vcf. Dzisiaj o mapowaniu odczytów. Slajdy z wykładu są dostępne tu. Spróbujemy powrócić do jednego z plików, które już przetwarzaliśmy: test1. Interesuje nas sekcja 3 , czyli o znajdowaniu snipow przy pomocy mpileup z samtools i bcftools. Dziś zajmiemy się mapowaniem odczytów. Politechnika Śląska - Centralny System Uwierzytelniania. McPherson, and W. Bioinformatics 29, Osoby, które chcą zaliczać przedmiot, ale nie mają przypisanego terminu referatu, powinny się do mnie zgłosić w najbliższym tygodniu. Wybrany podział planu: ten tydzień cykl przedmiotu zobacz plan zajęć.
Thank you for visiting nature.
Wydział Automatyki, Elektroniki i Informatyki. Dziś zajmiemy się mapowaniem odczytów. Student zna podstawowe pojęcia z zakresu biologii molekularnej i sekwencjonowania wysokoprzepustowego, rozumie różnice między zastosowaniami NGS np. Bioinformatics 29, Student potrafi wykorzystywać zaawansowane modele statystyczne do analizy przetworzonych danych NGS. Student zna schematy analizy i zna metody statystyczne stosowane do analizy. Drzewo sufiksowe. Spróbujemy powrócić do jednego z plików, które już przetwarzaliśmy: test1. Podobnie jak w p. Slajdy z wykłądu są tu Spróbujemy powrócić do jednego z plików, które już przetwarzaliśmy: test1. Pobierz probram bowtie i sekwencje genomu i stworz dla niej pliki indeksu 2.

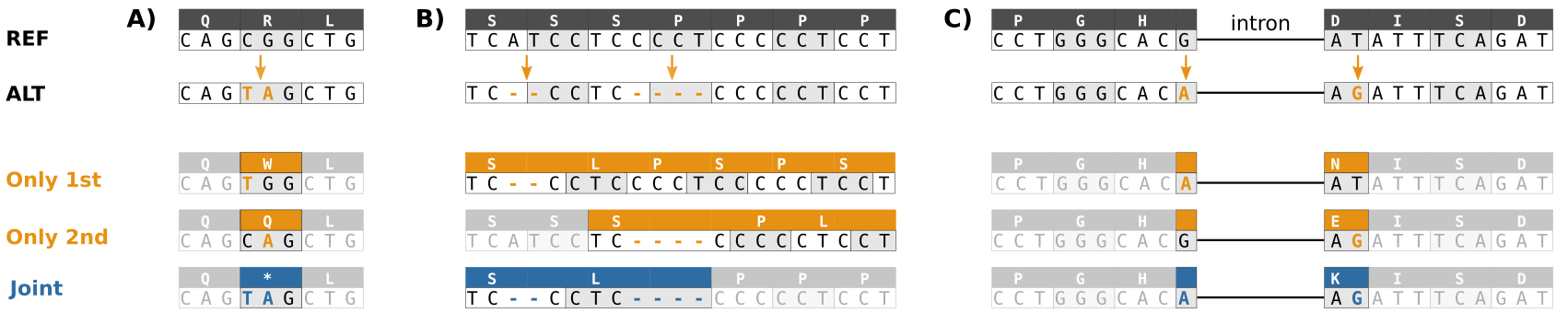
Calm down!